Base de datos de los metagenomas
PROYECTO PUNTERO. Investigadores de la ULE participan en la primera base de datos de metagenomas de alimentos. Contiene genomas de 3.600 especies microbianas diferentes, de las cuales 290 son especies nuevas y es de acceso abierto para su uso extensivo en estudios de microbiomas y aplicaciones en las industrias alimentarias.
Coral Barcenilla, Avelino Álvarez y José Cobo. DL
dl
La base de datos contiene genomas de 3.600 especies microbianas diferentes, de las cuales 290 son especies nuevas y es de acceso abierto para su uso extensivo en estudios de microbiomas y aplicaciones en las industrias alimentarias.
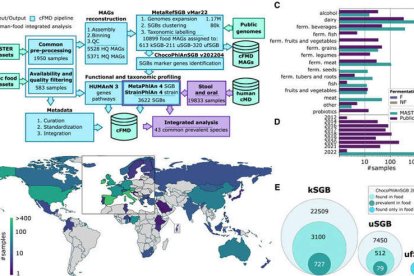
El proyecto Máster, financiado con fondos europeos y en el que han participado investigadores del área de Tecnología de los Alimentos de la Universidad de León (ULE), ha desarrollado la primera base de datos de metagenomas de alimentos (el término colectivo que designa todo el material genómico de todos los microorganismos de un entorno, en este caso los alimentos). El artículo científico donde se describe la creación de la base de datos, ‘Unexplored microbial diversity from 2,500 food metagenomes and links with the human microbiome’ (Diversidad microbiana inexplorada de 2.500 metagenomas de alimentos y vínculos con el microbioma humano), se ha publicado ayer, jueves 29 de agosto, en la prestigiosa revista Cell. Los microorganismos pueden tener un impacto positivo en la producción de alimentos, como por ejemplo a través de la fermentación de los mismos, o un impacto negativo cuando causan el deterioro del alimento o enfermedades de transmisión alimentaria. Hasta hace poco, el análisis de estos microbios se ha basado en gran medida en enfoques tradicionales que requerían el crecimiento de estos microorganismos en placas de Petri, con diferentes medios selectivos utilizados para el aislamiento de diferentes microorganismos.
La aplicación de métodos basados en técnicas de secuenciación de ADN de alto rendimiento, ha demostrado tener potencial para mejorar el análisis microbiológico de alimentos, ya que permite realizar pruebas rápidas y simultáneas de todos los microorganismos en paralelo, incluidos aquellos que son difíciles de cultivar. De esta forma, se consigue obtener información pormenorizada de una comunidad microbiana.
La creación de esta nueva base de datos ha sido posible gracias a la secuenciación de miles de muestras de alimentos recogidas durante el proyecto Máster en instalaciones de procesamiento de alimentos de toda Europa.
La existencia de la nueva base de datos significa que, en el futuro, los datos generados a partir del análisis basado en la secuenciación de ADN de muestras de alimentos se podrán analizar de forma rápida y precisa para identificar y controlar rápidamente los microorganismos indeseables, al tiempo que se mejorarán los atributos promotores de la salud de los alimentos que contienen microorganismos deseables. La base de datos es un recurso de acceso abierto, lo que facilitará la aplicación a gran escala de esta tecnología en todo el mundo por parte de académicos y de la industria alimentaria.
La base de datos contiene genomas de 3.600 especies microbianas diferentes, de las cuales 290 son especies nuevas. Está disponible de forma gratuita (https://github.com/SegataLab/cFMD) para su uso extensivo en estudios de microbiomas y aplicaciones en las industrias alimentarias. Por ejemplo, para conocer la dispersión de microorganismos a lo largo de la cadena alimentaria, estudiar la propagación de genes de resistencia a los antimicrobianos, detectar microorganismos indeseables o investigar la transmisión de microorganismos a los seres humanos. La disponibilidad de la base de datos representa un gran paso hacia un futuro en el que la secuenciación metagenómica podría complementar o sustituir a la microbiología clásica como una herramienta de análisis microbiológico en la cadena alimentaria más precisa y rápida.
Esta es la esencia de un trabajo de investigación puntero.

Coral Barcenilla, Avelino Álvarez y José Cobo. DL